Der Innovationsbereich Cross-funktionales Imaging bündelt mehrere Imaging Technologien zur Forschung und Diagnostik, die in präklinischen und klinischen Bereichen eingesetzt werden. Diese umfassen neben klassischen histologischen und immunhistologischen Methoden, auch modernste Imaging Technologien wie das nicht-invasive in vivo Fluoreszenz- und Lumineszenz-Imaging im Tiermodell, dem High-Content Imaging mittels ImageXpress, sowie der strahlungsfreien Untersuchung von Mikrozirkulationsstörungen der Hände (Xiralite-Plattform) zur im klinischen Bereich.
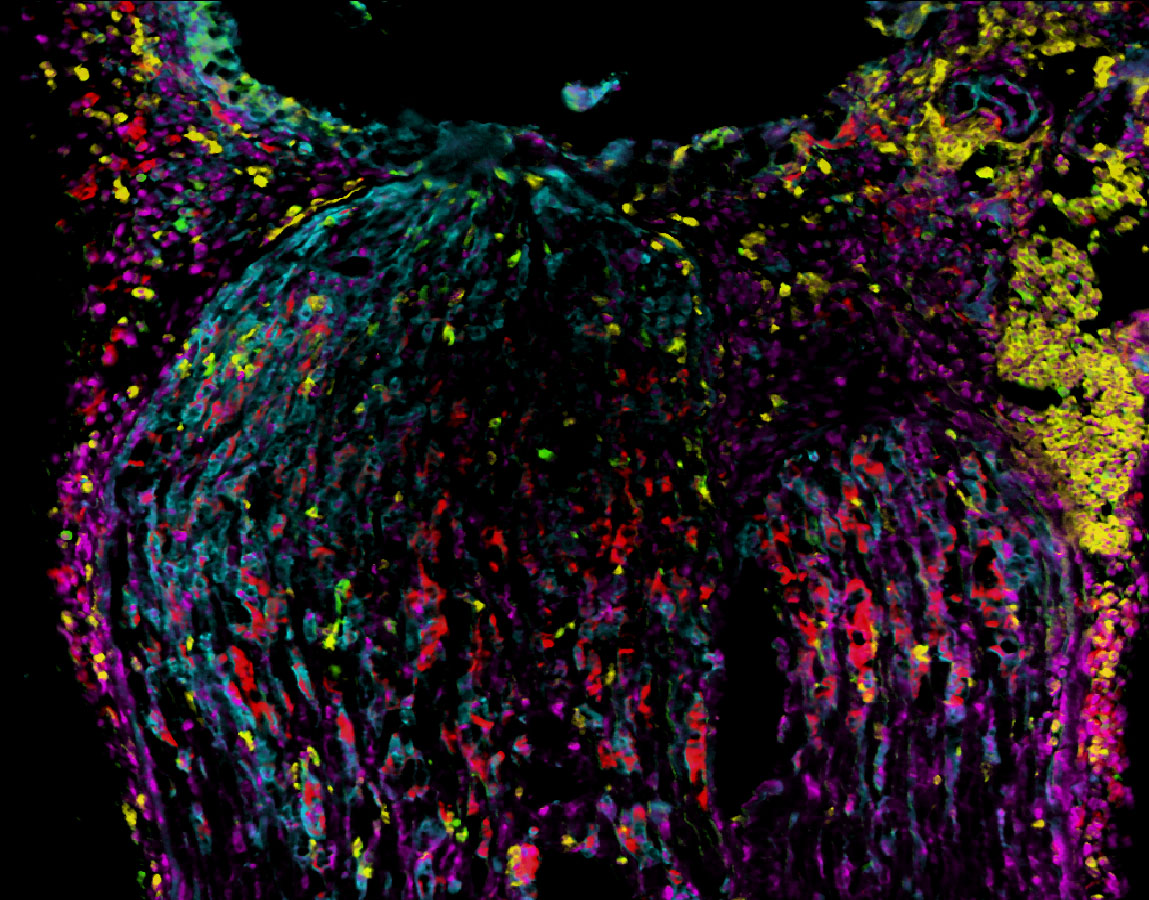
Kerntechnologie ist die »SMILE« (Sequentielle Multiple Immunhistochemie zur Lokalisations- und Expressionsanalyse) Technologie für das multiple sequentielle Immunhistologische Imaging für die Visualisierung von bis zu 100 Proteinen im gleichen histologischen Präparat, die für präklinische und klinische Proben zum Einsatz kommt. Durch innovative bioinformatische Analysen können zelluläre Nachbarschaften und Mikroumgebungen in den Präparaten quantitativ beschrieben werden. Dies ermöglicht u.a. eine hochsensible Analyse der Wirkungen von Wirkstoffen auf Immunantworten. Durch den Einsatz der großen Anzahl von Antikörpern wird der Informationsgewinn aus Gewebeproben optimiert und gleichzeitig die Menge an benötigtem Probenmaterial stark reduziert. Dies ist insbesondere bei Patienten- aber auch bei Tierproben relevant. Bei Patientenproben ergibt sich zudem die Möglichkeit schneller und preiswerter Diagnostik, sowie individualisierter Therapieempfehlungen.
Kernkompetenzen:
- SMILE System zur multiplen sequentiellen Immunhistochemie
- IVIS Spektrum zum in vivo Fluoreszenz- und Lumineszenz-Imaging
- High-Content Imaging mittels konfokaler Fluoreszenzmikroskopie (z. B. ImageXpress)
- Single cell Calcium Imaging
- Xiralite-Diagnostik zur strahlungsfreien Untersuchung von Mikrozirkulationsstörungen der Hände im klinischen Bereich
 Fraunhofer-Institut für Translationale Medizin und Pharmakologie ITMP
Fraunhofer-Institut für Translationale Medizin und Pharmakologie ITMP